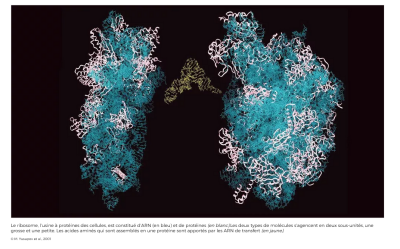
Des premières images à la reconstitution du phénomène
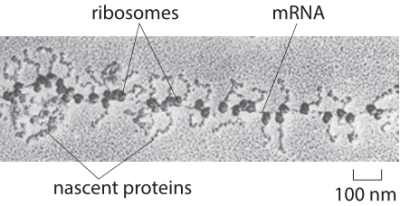
Les ribosomes ont été observés pour la première fois en 1938 par le biologiste belge Albert Claude. En étudiant le virus du sarcome de Rous, il détecte sous son microscope, en plus des virus, de petites particules présentes dans différents tissus : il les nomme microsomes. Près de 20 ans plus tard, grâce à des micrographies électroniques, le biologiste américain George Palade obtient une image plus claire de ces particules dont il estime la taille à environ 20 nanomètres. À la même époque, le biologiste américain Paul Zamecnik et ses collègues utilisent des acides aminés radioactifs, et montrent que les microsomes fabriquent des protéines. Leur composition riche en acide ribonucléique (arn) est établie en 1958 et conduit le biochimiste américain Richard Roberts à les nommer ribosomes.
La détermination de la structure tridimensionnelle des ribosomes a monopolisé les efforts des biologistes structuraux pendant plusieurs décennies. En 2000, la structure complète à l'échelle atomique semblait encore un « rêve » inaccessible.
Un an après l’attribution du prix Nobel de chimie pour la découverte de la structure atomique du ribosome bactérien, les chercheurs de l’Institut de génétique et de biologie moléculaire et cellulaire (CNRS/Université de Strasbourg/Inserm) ont déterminé la première structure d’un ribosome eucaryote, celui de la levure. Ces travaux publiés le 26 novembre 2010 dans la revue Science mettent fin à une course internationale effrénée pour la détermination de la structure de cette imposante machinerie cellulaire. Le ribosome eucaryote est actuellement la plus grande molécule asymétrique biologique dont la structure a été élucidée par cristallographie. Ces résultats ouvrent de nouvelles pistes de recherche pour la compréhension de la dynamique de la synthèse protéique et pour le développement de nouveaux composés thérapeutiques.
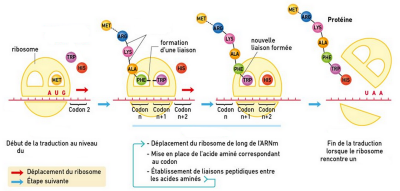
La première étape de la synthèse d’une protéine est celle de l’assemblage d’un intermédiaire porteur d'acide aminé (il s’agit toujours de celui qui porte l’acide aminé méthionine) et de l’ ARN messager à la petite sous-unité du ribosome. Ensuite, la grande sous-unité s’associe à ce complexe. Le ribosome ainsi constitué est doté de deux sites majeurs, où se « nichent » les molécules porteuses d'acides aminés. Dans le 1er site chaque fois que la petite sous-unité détecte une association convenable entre un porteur d'acide aminé et un codon de l’ARN messager, l’acide aminé est positionné dans une poche de la grande sous-unité. Dans ce centre, une liaison peptidique (qui unit les acides aminés) se forme entre l’acide aminé nouvellement arrivé et l’acide aminé méthionine.
À ce moment, le premier site est vide (il ne porte pas d’acides aminés), l’autre porte la chaîne constituée des deux acides aminés. Grâce à une série de mouvements coordonnés, le ribosome se déplace jusqu’au codon suivant de l’ARN messager. La molécule porteuse de la protéine en devenir passe du 1er au 2ème site et le processus se répète. La protéine synthétisée par addition successive d’acides aminés s’allonge et traverse la grande sous-unité du ribosome par une sorte de «canal», pour sortir à l’arrière du ribosome. Là, elle se replie à mesure de sa fabrication pour adopter sa structure spatiale fonctionnelle.
Quand le ribosome rencontre un codon dit Stop, les éléments du complexe se dissocient, libérant la protéine.
Chez la bactérie Escherichia coli, plus d’un million de liaisons peptidiques sont construites chaque seconde. Un ribosome seul relie entre 10 et 20 acides aminés par seconde et élabore une petite protéine (environ 150 acides aminés) en une minute et une grosse protéine, telle la titine du muscle (environ 25 000 acides aminés) en quelques heures.
Modifié d'après Pour la Science n°313, Le ribosome : l'usine à protéines.
Représenter la traduction
Question⚓
Modéliser la transcription et la traduction avec un programme Python
Question⚓
Il est possible de modéliser la transcription et la traduction en utilisant un programme écrit en langage Python.
Réalisez un tableau de comparaison entre le phénomène naturel et son déroulement avec le modèle numérique proposé.
Pour cela, analysez puis exécutez le programme Python de transcription et de traduction suivant : https://capytale2.ac-paris.fr/web/c/7e67-5476459.
Des éléments pour l'aide à l'analyse du code Python sont présents dans la partie solution ci-dessous.
Aide : identifiez ce qui reproduit correctement le phénomène naturel et ce qui s'en éloigne (par exemple pour la lecture de l'ARNm, la présence d'un ribosome...).
Solution⚓
- 1 Variables

Les variables sequence, complémentaire, transcrit et traduit sont définies. Les 3 dernières sont vides avant exécution du code.
- 2 Boucle for : répliquer la séquence

Exécution d'un bloc de code pour la variable nucléotide à l'intérieur de la suite de lettre de la variable sequence.
Utilisation de l'opérateur += qui permet d'incrémenter (ajouter) un éléments dans la variable.
Instruction conditionnelle if :
Au cours de la boucle pour le 1er nucléotide si il est égal à A alors la variable complémentaire se remplit avec la lettre T.
Instruction conditionnelle elif : (else if) sinon le programme teste si ce nucléotide est T et si c'est le cas alors la variable complémentaire se remplit avec la lettre A.
Instruction conditionnelle elif : (else if) sinon le programme teste si ce nucléotide est G et si c'est le cas alors la variable complémentaire se remplit avec la lettre C.
Instruction conditionnelle elif : (else if) sinon le programme teste si ce nucléotide est C et si c'est le cas alors la variable complémentaire se remplit avec la lettre G.
La fonction print( ) permet d'afficher un texte entre guillemets et le contenu de la variable.
- 3 Boucle for : transcrire la séquence

Il s'agit du même principe mais avec une complémentarité de nucléotides différente.

- 1 Variables

Déclaration des variables numéro et numbis d'abord affectées de la valeur 0.
Les variables qui contiennent les caractères qui correspondent aux nucléotides sont indicées et le premier nucléotide de la liste possède l'indice 0.
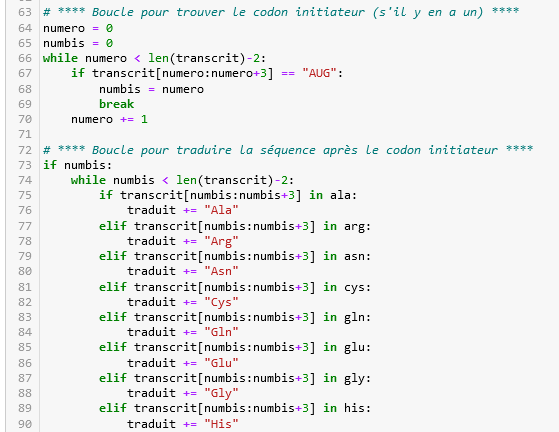
- 2 Trouver le codon d'initiation dans l'ARNm

la fonction len renvoie la longueur (nombre d'éléments) d'un objet.
La boucle conditionnelle while correspond à « tant que ».
La fonction s'exécute tant que le nombre d'éléments dans transcrit -2 est plus grand que numero et jusqu'à ce que cette condition vérifie si la sous-chaîne de transcrit qui commence à l'index numero et qui a une longueur de 3 caractères est égale à "AUG".
Si cette condition se vérifie l'index de numéro est stocké dans numbis et la boucle stoppe (break).
Sinon on affecte à la variable numero une valeur augmentée de 1 et on teste la position suivante dans la chaîne.
- 3 Traduction de l'ARNm à partir du codon d'initiation
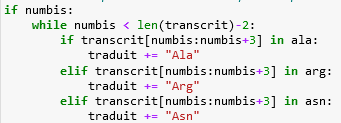
numbis correspond maintenant à l'indice de la première lettre du deuxième codon après AUG
Ces conditions vérifient si la sous-chaîne de 3 caractères, à partir de l'index numbis, est présente dans la liste ala qui contient les codons correspondant à cet acide aminé. Si l'un des codon est trouvé Ala est ajoutée à la variable « traduit ».
Si ce n'est pas le cas les codons de l'acide aminé suivant stockés dans la liste correspondante sont testés jusqu'à (while = tant que) ce qu'un acide aminé soit trouvé.
Quand il est trouvé, la variable numbis est incrémentée de 3. Cela signifie que le code passe à la prochaine suite de 3 caractères dans la variable « transcrit ».

Si le codon trouvé correspond à un codon "Stop", Stop est ajouté à « traduit » et la boucle est interrompue avec break. Cela signifie que si une séquence de 3 caractères correspondant à "stop" est trouvée, le processus de traduction s'arrête immédiatement.